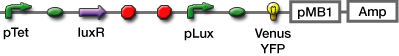Team:Chiba/Reporter
From 2008.igem.org
(New page: <html><link rel="stylesheet" href="https://2008.igem.org/wiki/index.php?title=User:Maiko/chiba.css&action=raw&ctype=text/css" type="text/css" /></html> Image:Chiba-U.gif {| style="color...) |
|||
| (49 intermediate revisions not shown) | |||
| Line 13: | Line 13: | ||
==Reporter== | ==Reporter== | ||
| - | + | 本プロジェクトではある一定時間がたった時に、遺伝子発現が起こるタイマーを作ることが目的 | |
| - | == | + | レシーバーの遺伝子発現から確認までのタイムラグができるだけ少ない出力が必要 |
| - | * | + | |
| - | * | + | そこで候補となったのは、以下の3種類 |
| + | *Fluorescent Protein | ||
| + | *Bioluminescence(luciferase) | ||
| + | *Cell staining (LacZ and X-gal assay) | ||
| + | |||
| + | |||
| + | 蛍光たんぱくとβ-gal(lacZ)を実験で使用した。 | ||
| + | |||
| + | |||
| + | 蛍光たんぱくとしてGFP、maturationの早いVenus YFP[http://www.nature.com/nbt/journal/v20/n1/full/nbt0102-87.html|(1)],目視での確認が容易なmCherry[http://www.ncbi.nlm.nih.gov/pubmed/15558047|(2)],を選んだ。 | ||
| + | |||
| + | Luciferaseは化学発光であるために、発現の確認には暗所でなければならない。使用条件が限られてしまうため候補から外した | ||
| + | |||
| + | ==Experiment== | ||
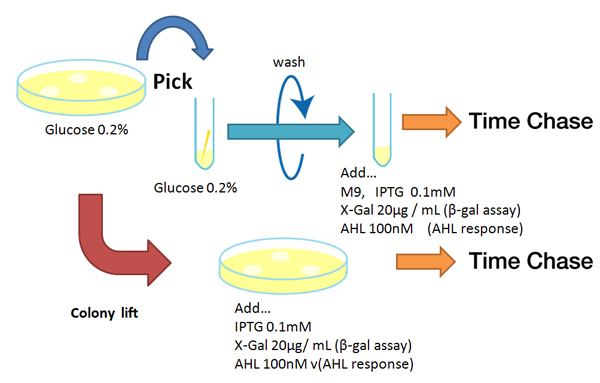
| + | [[Image:Reporter method Chiba.jpg|rifht|thumb|'''Fig. '''レポーターのタイムレスポンスの実験方法]] | ||
| + | Strain:XL10G Kan<sup>R</sup> | ||
| + | |||
| + | *Fluorescent Protein | ||
| + | :*GFP | ||
| + | ::*pGFPuv | ||
| + | ::*[http://partsregistry.org/Part:BBa_T9002 BBa_T9002] | ||
| + | ::[[Image:Reporter 9002 chiba.gif]] | ||
| + | :*Venus YFP | ||
| + | ::*[http://partsregistry.org/wiki/index.php?title=Part:BBa_K084003 BBa_K084003] | ||
| + | ::[[Image:K084003 chiba.gif]] | ||
| + | ::*pLac-Venus YFP | ||
| + | ::[[Image:pLac_Venus_Chiba.gif]] | ||
| + | :*mCherry | ||
| + | ::*pLac-mCherry | ||
| + | ::[[Image:PLac_mChe_Chiba.gif]] | ||
*β-gal (X-gal assay) | *β-gal (X-gal assay) | ||
| + | :*PUC19(plac-LacZα) | ||
| + | ::[[PLac-lacZ-Chiba.gif]] | ||
| + | |||
| + | *Equipment | ||
| + | :shaking incubator | ||
| + | ::Innova 4200 Benchtop or Floor-Stackable Incubator Shaker(37°C) | ||
| + | :46-well plate(deep well) | ||
| + | :96-well plate(deep well) | ||
| + | :Fluoroskan Ascent 2.5(program:Ascent Software Version 2.6) | ||
| + | :Beckman Allegratm X-12R Centrifuga(Beckman Coulter) | ||
| + | |||
| + | |||
| + | |||
| + | *固体編 | ||
| + | 固体培地(pLac:0.2%グルコース入り)にまく<br> | ||
| + | コロニーを新しい固体培地(pLac:IPTG, plux:AHL, LacZ:IPTG&X-gal入り)にコロニーリフトする,計測開始<br> | ||
| + | 37°Cのしんとう培養器で培養<br> | ||
| + | 30分ごとに目視で確認できるか観察<br> | ||
| + | |||
| + | *液体編 | ||
| + | プレ培<br> | ||
| + | コロニーを固体培地からピックし,LB培養液2ml(pLacであれば0.2%グルコース入り)で12時間培養(37°C)<br> | ||
| + | 本培<br> | ||
| + | OD値を計測して、菌数を合わせるため培養液を1/200に希釈する。<br> | ||
| + | OD=1.00になるまで37°Cで培養<br> | ||
| + | 生理食塩水で洗浄(2.0min 8,000rpm)x2<br> | ||
| + | 46穴または96穴deep wellにM9(2ml)<br> | ||
| + | IPTG(最終濃度0.1mM)またはAHL(最終濃度100nM)を加えて、計測開始<br> | ||
| + | 30分ごとに蛍光強度、OD値を計測、また目視で確認できるか観察<br> | ||
| + | |||
| + | ==Result & Discussion== | ||
| + | '''Result''' | ||
| + | '''LacZ''' | ||
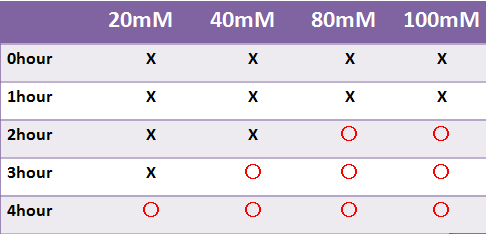
| + | [[Image:LacZ chiba.gif|thumb|right|'''Table.''' X-gal濃度を変化させ、染色の様子を測定した結果]] | ||
| + | *蛍光たんぱくではないLacZは目視のみで発現を確認した | ||
| + | *LacZはX-galの濃度によって目視での確認時間が変わった | ||
| + | *液体で4時間以内に目視で確認することはできなかった | ||
| + | ::-->8時間後目視で確認できた | ||
| + | |||
| + | '''蛍光たんぱく''' | ||
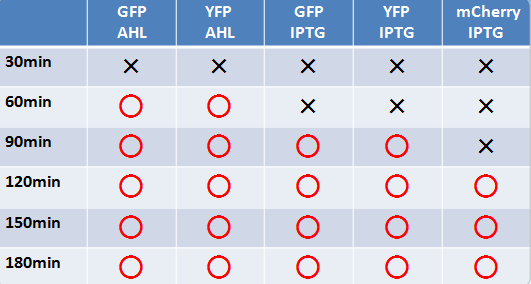
| + | [[Image:Reporter chiba.gif|thumb|right|'''Table.''' それぞれのレポーター遺伝子の評価をした結果]] | ||
| + | *液体、固体培地において似たような結果になった | ||
| + | *目視で一番見やさはmCherry,GFP,YFPの順であった | ||
| + | *固体培地のほうが目視で確認しやすい | ||
| + | *AHLでのインダクションのほうがIPTGより確認できた時間が早かった | ||
| + | *GFP,YFPのほうがmCherryより確認できた時間が早かった | ||
| + | |||
| + | |||
| + | <br clear=all> | ||
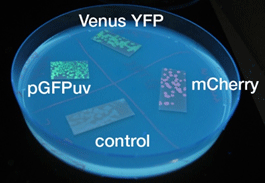
| + | [[Image:Soliduv Chiba.gif|thumb|right|'''Fig'''UVを当てた固体培地上のレポーター]] | ||
| + | |||
| + | '''Discussion''' | ||
| + | |||
| + | このプロジェクトでは、発現するまでの時間を変えることが目的である | ||
| + | |||
| + | *X-galの濃度を高くすると発現を確認できるまでの時間は短くなったが、蛍光たんぱくで発現を確認できるまでの時間より遅い | ||
| + | |||
| + | *X-galを基質とするβ-galでは、時間がたつごとに基質濃度が下がり、出力までの時間が長くなってしまうことも考えられる | ||
| + | |||
| + | 以上の2つの理由から蛍光タンパク質が適している | ||
| + | |||
| + | またGFP,YFPの発現時間までのタイムラグが一番少なかった | ||
| + | |||
| + | *目視で確認した際、GFPのほうがYFPよりも目視で確認するのが容易であった | ||
| + | 以上の理由から、出力はGFPとする。 | ||
| + | <br clear=all> | ||
{| style="color:white;background-color:Maroon" cellpadding="3" cellspacing="3" border="1" bordercolor="white" width="100%" align="center" | {| style="color:white;background-color:Maroon" cellpadding="3" cellspacing="3" border="1" bordercolor="white" width="100%" align="center" | ||
!align="center"|[[Team:Chiba|Home]] | !align="center"|[[Team:Chiba|Home]] | ||
Latest revision as of 19:42, 29 October 2008
| Home | The Team | The Project | Parts Submitted to the Registry | Reference | Notebook | Acknowledgements |
|---|
Reporter
本プロジェクトではある一定時間がたった時に、遺伝子発現が起こるタイマーを作ることが目的
レシーバーの遺伝子発現から確認までのタイムラグができるだけ少ない出力が必要
そこで候補となったのは、以下の3種類
- Fluorescent Protein
- Bioluminescence(luciferase)
- Cell staining (LacZ and X-gal assay)
蛍光たんぱくとβ-gal(lacZ)を実験で使用した。
蛍光たんぱくとしてGFP、maturationの早いVenus YFP[http://www.nature.com/nbt/journal/v20/n1/full/nbt0102-87.html|(1)],目視での確認が容易なmCherry[http://www.ncbi.nlm.nih.gov/pubmed/15558047|(2)],を選んだ。
Luciferaseは化学発光であるために、発現の確認には暗所でなければならない。使用条件が限られてしまうため候補から外した
Experiment
Strain:XL10G KanR
- Fluorescent Protein
- GFP
- Venus YFP
- mCherry
- β-gal (X-gal assay)
- PUC19(plac-LacZα)
- Equipment
- shaking incubator
- Innova 4200 Benchtop or Floor-Stackable Incubator Shaker(37°C)
- 46-well plate(deep well)
- 96-well plate(deep well)
- Fluoroskan Ascent 2.5(program:Ascent Software Version 2.6)
- Beckman Allegratm X-12R Centrifuga(Beckman Coulter)
- 固体編
固体培地(pLac:0.2%グルコース入り)にまく
コロニーを新しい固体培地(pLac:IPTG, plux:AHL, LacZ:IPTG&X-gal入り)にコロニーリフトする,計測開始
37°Cのしんとう培養器で培養
30分ごとに目視で確認できるか観察
- 液体編
プレ培
コロニーを固体培地からピックし,LB培養液2ml(pLacであれば0.2%グルコース入り)で12時間培養(37°C)
本培
OD値を計測して、菌数を合わせるため培養液を1/200に希釈する。
OD=1.00になるまで37°Cで培養
生理食塩水で洗浄(2.0min 8,000rpm)x2
46穴または96穴deep wellにM9(2ml)
IPTG(最終濃度0.1mM)またはAHL(最終濃度100nM)を加えて、計測開始
30分ごとに蛍光強度、OD値を計測、また目視で確認できるか観察
Result & Discussion
Result LacZ
- 蛍光たんぱくではないLacZは目視のみで発現を確認した
- LacZはX-galの濃度によって目視での確認時間が変わった
- 液体で4時間以内に目視で確認することはできなかった
- -->8時間後目視で確認できた
蛍光たんぱく
- 液体、固体培地において似たような結果になった
- 目視で一番見やさはmCherry,GFP,YFPの順であった
- 固体培地のほうが目視で確認しやすい
- AHLでのインダクションのほうがIPTGより確認できた時間が早かった
- GFP,YFPのほうがmCherryより確認できた時間が早かった
Discussion
このプロジェクトでは、発現するまでの時間を変えることが目的である
- X-galの濃度を高くすると発現を確認できるまでの時間は短くなったが、蛍光たんぱくで発現を確認できるまでの時間より遅い
- X-galを基質とするβ-galでは、時間がたつごとに基質濃度が下がり、出力までの時間が長くなってしまうことも考えられる
以上の2つの理由から蛍光タンパク質が適している
またGFP,YFPの発現時間までのタイムラグが一番少なかった
- 目視で確認した際、GFPのほうがYFPよりも目視で確認するのが容易であった
以上の理由から、出力はGFPとする。
| Home | The Team | The Project | Parts Submitted to the Registry | Reference | Notebook | Acknowledgements |
|---|
 "
"