Team:Chiba/AHL Receiver Phase
From 2008.igem.org
(→Result) |
(→Method) |
||
| (31 intermediate revisions not shown) | |||
| Line 11: | Line 11: | ||
|} | |} | ||
__NOTOC__ | __NOTOC__ | ||
| - | + | ||
==Quorum Sensing Crosstalk== | ==Quorum Sensing Crosstalk== | ||
===Design=== | ===Design=== | ||
| - | [[Image:Table R chiba.gif|frame|left|Table. | + | [[Image:Table R chiba.gif|frame|left|'''Table.''' LuxR family gene '''Data modified from M.RIVAS et.al. Biol Res 38: 283-297, (2005)''']] |
異なる生物はそれぞれに異なるLuxRタイプのタンパク質を持ち、それぞれアシル鎖の長さ、あるいはC-3位の置換基が異なる種類のAHLに応答する。生物種によって異なるAHLと、それに応答するLuxRタイプのタンパク質は以下の表のよう。 | 異なる生物はそれぞれに異なるLuxRタイプのタンパク質を持ち、それぞれアシル鎖の長さ、あるいはC-3位の置換基が異なる種類のAHLに応答する。生物種によって異なるAHLと、それに応答するLuxRタイプのタンパク質は以下の表のよう。 | ||
Pseudomonas aeruginosa由来のRhlR,LasRも3OC6HSLを受け取ることがわかっている。 | Pseudomonas aeruginosa由来のRhlR,LasRも3OC6HSLを受け取ることがわかっている。 | ||
<br clear=all> | <br clear=all> | ||
| - | [[Image:Lux box Chiba.jpg|frame| | + | [[Image:Lux box Chiba.jpg|frame|left|'''Fig.''' Comparison of the predicted Lux box. '''Data modified from A.Fekete et.al. Anal Bioanal Chem 387:455–467,(2007)''']] |
| - | また、LuxR protein familyはLux boxという特定の配列を持ったプロモーター下にある遺伝子の発現を活性化する。 | + | また、LuxR protein familyはLux boxという特定の配列を持ったプロモーター下にある遺伝子の発現を活性化する。(Fig. ) |
Lux boxも生物種によって配列が異なるが、ほとんど変わらない配列を持っているために違った生物種のLuxR protein familyであっても、AHLが存在下で遺伝子発現を活性化すると考えられる。 | Lux boxも生物種によって配列が異なるが、ほとんど変わらない配列を持っているために違った生物種のLuxR protein familyであっても、AHLが存在下で遺伝子発現を活性化すると考えられる。 | ||
| Line 46: | Line 46: | ||
[[Image:LuxI-sender Chiba.gif|left|]]<br clear=all> | [[Image:LuxI-sender Chiba.gif|left|]]<br clear=all> | ||
| - | *''' | + | *'''Transcriptional regulator''' |
| - | + | :*LuxR | |
[[Image:LuxR-p15A Chiba.gif]] | [[Image:LuxR-p15A Chiba.gif]] | ||
| - | + | :*RhlR[http://partsregistry.org/Part:BBa_K084004 (BBa_K084004)] | |
[[Image:RhlR-p15A Chiba.gif]] | [[Image:RhlR-p15A Chiba.gif]] | ||
| - | + | :*LasR[http://partsregistry.org/Part:BBa_K084005 (BBa_K084005)] | |
[[Image:LasR-p15A Chiba.gif]] | [[Image:LasR-p15A Chiba.gif]] | ||
| - | + | :*CinR[http://partsregistry.org/Part:BBa_K084006 (BBa_K084006)] | |
[[Image:CinR-p15A Chiba.gif]] | [[Image:CinR-p15A Chiba.gif]] | ||
*'''Reporter''' | *'''Reporter''' | ||
| - | + | :*Plux-GFP[http://partsregistry.org/Part:BBa_J37032 (BBa_J37032)] | |
[[Image:Plux-GFP-pMB1 Chiba.gif]] | [[Image:Plux-GFP-pMB1 Chiba.gif]] | ||
| - | |||
| - | |||
===Result & Discussion=== | ===Result & Discussion=== | ||
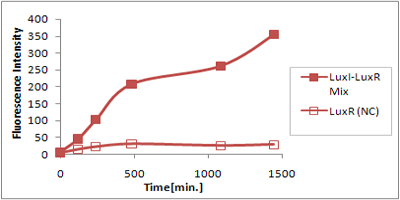
| - | [[Image:R-family-Crosstalk-Lux Chiba.gif|thumb|left|'''Fig.'''Time Delay Test LuxI | + | [[Image:R-family-Crosstalk-Lux Chiba.gif|thumb|left|'''Fig.'''Time Delay Test Sender:LuxI,Receiver:LuxR]] |
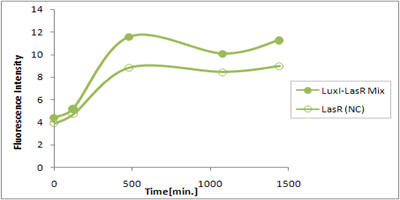
| - | [[Image:R-family-Crosstalk-Las Chiba.gif|thumb|left|'''Fig.'''Time Delay Test LuxI | + | [[Image:R-family-Crosstalk-Las Chiba.gif|thumb|left|'''Fig.'''Time Delay Test Sender:LuxI, Receiver:LasR]] |
| - | [[Image:R-family-Crosstalk-Rhl Chiba.gif|thumb|left|'''Fig.'''Time Delay Test LuxI | + | [[Image:R-family-Crosstalk-Rhl Chiba.gif|thumb|left|'''Fig.'''Time Delay Test Sender:LuxI, Receiver:RhlR]] |
<br clear=all> | <br clear=all> | ||
| Line 75: | Line 73: | ||
LuxRを発現させたとき、GFPは多く発現していることが分かる(Fig.1)が、そのほかのLasR,RhlRを発現させた場合、ほとんど蛍光強度が上昇していない。(Fig.2,Fig.3) | LuxRを発現させたとき、GFPは多く発現していることが分かる(Fig.1)が、そのほかのLasR,RhlRを発現させた場合、ほとんど蛍光強度が上昇していない。(Fig.2,Fig.3) | ||
つまり、遺伝子発現が活性化されていることが確認できなかった。 | つまり、遺伝子発現が活性化されていることが確認できなかった。 | ||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
クロストークさせると、発現量が大きく減少してしまうのでタイムラグを確認できるほど遺伝子が発現がGFPでは確認できなかった | クロストークさせると、発現量が大きく減少してしまうのでタイムラグを確認できるほど遺伝子が発現がGFPでは確認できなかった | ||
| Line 85: | Line 78: | ||
LuxI->LasR->PLas,LuxI->RhlR->PRhlは元来発現強度が弱いので[http://www3.interscience.wiley.com/journal/119124142/abstract (1)]、LVAの影響により、さらに発現が見えにくくなったと考えられる。 | LuxI->LasR->PLas,LuxI->RhlR->PRhlは元来発現強度が弱いので[http://www3.interscience.wiley.com/journal/119124142/abstract (1)]、LVAの影響により、さらに発現が見えにくくなったと考えられる。 | ||
| - | + | よってLasRとRhlRのLVAをとり、Sender:LuxI, Receiver:LuxR,LasR,RhlRのクロストーク評価を行う必要がある。 | |
| + | |||
| Line 92: | Line 86: | ||
==Plasmid Copy number== | ==Plasmid Copy number== | ||
===Design=== | ===Design=== | ||
| - | + | レシーバー側のプラスミドのコピーナンバーを変えることで、遺伝子発現に時間差をつけることができると予想した。 | |
===Experiment=== | ===Experiment=== | ||
| - | + | oriがcolE1のハイコピーベクターにのったBBa_T9002と、oriがP15Aのローコピーベクターに乗ったT9002バリエーションを使用した。 | |
これらを比較することでコピーナンバーを変えることでの影響を調べた。 | これらを比較することでコピーナンバーを変えることでの影響を調べた。 | ||
| Line 112: | Line 106: | ||
これらのほかにBBa_T9002にはChloramphenicol耐性マーカーを持つ空ベクター、Low copyバリエーションにはAmpicillin耐性マーカーの空ベクターをダブルトランスフォーメーションさせた。 | これらのほかにBBa_T9002にはChloramphenicol耐性マーカーを持つ空ベクター、Low copyバリエーションにはAmpicillin耐性マーカーの空ベクターをダブルトランスフォーメーションさせた。 | ||
| - | === | + | ===Method=== |
| - | + | #Transformed Sender into E.coli strains(BWΔFliC) and Receivers into E.coli strain(BWΔFliC). | |
| - | + | #Inoculated them independently in liquid media. Incubated at 37c°; 12h. | |
| + | #Inoculated again at 37c°; upto about OD600=2.0 | ||
| + | #Washed them. | ||
| + | #Mixed them (Sender:Receiver=1000μl:1000μl). | ||
| + | #Incubated at 30°C. | ||
| + | #Measured intensity of green fluorescence at regular time intervals. | ||
<br clear=all> | <br clear=all> | ||
| - | |||
| - | |||
| - | |||
| - | == | + | == LuxR mutant (Under Plannning) == |
===Design=== | ===Design=== | ||
| - | |||
[[Image:Mutant-LuxR Chiba.gif|frame|right|'''Table.1''' Seisitivity of LuxR mutants. Data modified from Collins et.al. Mol. Microbiol. 55, 712–723 (2005)]] | [[Image:Mutant-LuxR Chiba.gif|frame|right|'''Table.1''' Seisitivity of LuxR mutants. Data modified from Collins et.al. Mol. Microbiol. 55, 712–723 (2005)]] | ||
| + | [[Image:AHL-activation-of-mutagenizenized LuxR molecules Chiba.gif|frame|right|'''Table.Ahl activation of mutagenized LuxR molecules. Data modfied from B.Kock et al. Microbiology (2005), 151, 3589-3602''']] | ||
| + | Collins et.al. described the hyper-sensitive variants of luxR to AHL.(1) | ||
| + | CollonsらとKockらによって、LuxRに変異を入れることでLuxRのAHLに対する感受性を変えることができると報告されている。<sup>(1),(2)</sup> | ||
| - | + | LuxRのAHL認識部位に変異を入れることで、AHLに対する感受性が変わる。 | |
| - | + | #Ile45PheとするとAHLに対する感受性が約10倍上昇する<sup>(1)</sup> | |
| - | ( | + | #Leu42AlaとするとAHLに対する感受性が1/15に減少する<sup>(2)</sup> |
| + | #Leu42SerとするとAHLに対する感受性が1/1000に減少する<sup>(2)</sup> | ||
| + | <br clear=all> | ||
| - | + | ===Reference=== | |
| + | #Collins et.al. Directed evolution of Vibrio fischeri LuxR for increased sensitivity to a broad spectrum of acyl-homoserine lactones. Mol. Microbiol. 55, 712–723 (2005) | ||
| + | #B.Koch et.al.:The LuxR receptor: the sites of interaction with quorum-sensing signals and inhibitors. Microbiol. 2005 Nov;151(Pt 11):3589-602. | ||
Latest revision as of 21:32, 29 October 2008
| Home | The Team | The Project | Parts Submitted to the Registry | Reference | Notebook | Acknowledgements |
|---|
Quorum Sensing Crosstalk
Design
異なる生物はそれぞれに異なるLuxRタイプのタンパク質を持ち、それぞれアシル鎖の長さ、あるいはC-3位の置換基が異なる種類のAHLに応答する。生物種によって異なるAHLと、それに応答するLuxRタイプのタンパク質は以下の表のよう。
Pseudomonas aeruginosa由来のRhlR,LasRも3OC6HSLを受け取ることがわかっている。
また、LuxR protein familyはLux boxという特定の配列を持ったプロモーター下にある遺伝子の発現を活性化する。(Fig. ) Lux boxも生物種によって配列が異なるが、ほとんど変わらない配列を持っているために違った生物種のLuxR protein familyであっても、AHLが存在下で遺伝子発現を活性化すると考えられる。
Experiment
LuxR protein familyを変えてクロストークさせる実験を行った。
AHL senderとしてVibrio fischeri由来のLuxIを常に発現させ、3OC6HSLを合成させる。
AHL ReceiverとしてLuxR protein familyを発現させるReceiver plasmidと,Lux promoter下にGFPを含むレポータープラスミドをダブルトランスフォーメーションさせた。
AHL senderによって合成されたAHLをAHL receiverが受取り、Lux promoter下のGFPが発現する。
蛍光強度を蛍光リーダーを使ってGFPの発現を計測し、遺伝子発現の活性を調べた。
使った遺伝子回路は以下の通りある。
- Sender
- AHL autoinucer[http://partsregistry.org/Part:BBa_S03623 (BBa_S03623) ]
- Transcriptional regulator
- LuxR
- RhlR[http://partsregistry.org/Part:BBa_K084004 (BBa_K084004)]
- LasR[http://partsregistry.org/Part:BBa_K084005 (BBa_K084005)]
- CinR[http://partsregistry.org/Part:BBa_K084006 (BBa_K084006)]
- Reporter
- Plux-GFP[http://partsregistry.org/Part:BBa_J37032 (BBa_J37032)]
Result & Discussion
さまざまなLux protein familyを発現させ、GFPの蛍光強度を測定した結果が左のグラフ
LuxRを発現させたとき、GFPは多く発現していることが分かる(Fig.1)が、そのほかのLasR,RhlRを発現させた場合、ほとんど蛍光強度が上昇していない。(Fig.2,Fig.3) つまり、遺伝子発現が活性化されていることが確認できなかった。 クロストークさせると、発現量が大きく減少してしまうのでタイムラグを確認できるほど遺伝子が発現がGFPでは確認できなかった
本実験では、LuxR以外のR protein familyには分解タグである,LVA tagがついていた。 LuxI->LasR->PLas,LuxI->RhlR->PRhlは元来発現強度が弱いので[http://www3.interscience.wiley.com/journal/119124142/abstract (1)]、LVAの影響により、さらに発現が見えにくくなったと考えられる。
よってLasRとRhlRのLVAをとり、Sender:LuxI, Receiver:LuxR,LasR,RhlRのクロストーク評価を行う必要がある。
Plasmid Copy number
Design
レシーバー側のプラスミドのコピーナンバーを変えることで、遺伝子発現に時間差をつけることができると予想した。
Experiment
oriがcolE1のハイコピーベクターにのったBBa_T9002と、oriがP15Aのローコピーベクターに乗ったT9002バリエーションを使用した。
これらを比較することでコピーナンバーを変えることでの影響を調べた。
以下の遺伝子回路を使って、実験を行った。
Sender
- AHL autoinucer[http://partsregistry.org/Part:BBa_S03623 (BBa_S03623) ]
Receiver
- [http://partsregistry.org/Part:BBa_T9002 BBa_T9002 (Express GFP in response to AHL)]
- Low Copy Receiver
これらのほかにBBa_T9002にはChloramphenicol耐性マーカーを持つ空ベクター、Low copyバリエーションにはAmpicillin耐性マーカーの空ベクターをダブルトランスフォーメーションさせた。
Method
- Transformed Sender into E.coli strains(BWΔFliC) and Receivers into E.coli strain(BWΔFliC).
- Inoculated them independently in liquid media. Incubated at 37c°; 12h.
- Inoculated again at 37c°; upto about OD600=2.0
- Washed them.
- Mixed them (Sender:Receiver=1000μl:1000μl).
- Incubated at 30°C.
- Measured intensity of green fluorescence at regular time intervals.
LuxR mutant (Under Plannning)
Design
Collins et.al. described the hyper-sensitive variants of luxR to AHL.(1) CollonsらとKockらによって、LuxRに変異を入れることでLuxRのAHLに対する感受性を変えることができると報告されている。(1),(2)
LuxRのAHL認識部位に変異を入れることで、AHLに対する感受性が変わる。
- Ile45PheとするとAHLに対する感受性が約10倍上昇する(1)
- Leu42AlaとするとAHLに対する感受性が1/15に減少する(2)
- Leu42SerとするとAHLに対する感受性が1/1000に減少する(2)
Reference
- Collins et.al. Directed evolution of Vibrio fischeri LuxR for increased sensitivity to a broad spectrum of acyl-homoserine lactones. Mol. Microbiol. 55, 712–723 (2005)
- B.Koch et.al.:The LuxR receptor: the sites of interaction with quorum-sensing signals and inhibitors. Microbiol. 2005 Nov;151(Pt 11):3589-602.
| Home | The Team | The Project | Parts Submitted to the Registry | Reference | Notebook | Acknowledgements |
|---|
 "
"