Team:Chiba/Project/j
From 2008.igem.org
| ホーム | メンバー紹介 | プロジェクト紹介 | 作成したDNAパーツ | ノート |
|---|
より詳しいプロジェクト内容はこちらのページに英語で記載されています。
Contents |
プロジェクトデザイン
[http://en.wikipedia.org/wiki/Water_clock Water clock-wikipedia.en 水時計]のような働きをする大腸菌をデザインした。Fig.3にその機構を示す。
1. センダーは、シグナル分子をゆっくりと一定の速度で合成する。シグナル分子は代謝されない(あるいは現実的な時間スケールであれば事実上代謝されない)ために、ゆっくりと時間をかけて(線形に)蓄積される。
2. レシーバーはシグナル分子の濃度が閾値に達したときに初めて、シグナル分子を検知し遺伝子スイッチをオンにする。異なるシグナル分子の場合、遺伝子スイッチは異なる速度で活性化される。システム全体は順番を持って起動するスイッチとして働く。
3.レシーバーの感度を変える、またはシグナル分子の蓄積速度を変えることで、スイッチの遅延時間を変えることができる。いくつかの遅延時間を持ったスイッチを使うことで、異なる多数の細胞機能を、順番に活性化することができる.
プロジェクトデザイン
Quorum Sensing
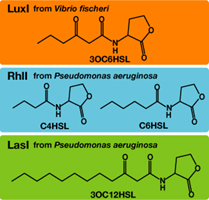
多くのグラム陰性細菌は、クオラムセンシングという機構という細胞間コミュニケーションシステムを用いて、同種の細胞の密度を知り、集団全体で協調的な遺伝子発現をする。このシステムは、N-アシルホモセリンラクトン(AHL)という分子を、シグナルとして使用する。AHLは"Iタンパク質"によって合成され、それが一定濃度を越えたとき、転写活性化タンパク質である"Rタンパク質"と結合し、遺伝子発現を誘導する。
感度差による応答時間の差
delay-timeをもった遺伝子発現を実現するために、バクテリア間の細胞間通信、クオラムセンシングを利用する。クオラムセンシングでは、応答閾値を超えるAHLが蓄積されたとき初めて、目的遺伝子の発現がおこる。AHL情報がじゅうぶんゆっくり蓄積するとき、ひとつのAHL送信装置に対して、感度の異なる複数の受信装置によってさまざまな出力装置を独立に起動すれば、感度の高いAHL 受信機ほど先に起動する。
Controlling the Delay time of the AHL signaling
LuxIのつくるAHLに対して、感度差による応答時間差は、2つの方法で実現することができる。
- LuxR/ Pluxの感度変異体を用いる
LuxRの変異体を用いれば、pluxがonになるAHL濃度が<1nMから~10nMまで変化する。[http://authors.library.caltech.edu/5553/ C. H. Collins.et al.Mol.Microbiol.2005.55(3).712–723] - クロストークを使う(その1)
AHL=Acyl-homoserine Lactoneには、そのアシル基が異なる種類がある。luxIが合成するAHLは、3-oxo-hyxanoyl-Homoserine Lactone (=OHHL)であり、3-oxo-dodecanoyl-homoserine Lactone(=ODHL)や、butyryl-homoserine Lactone(=HHL)が存在する。それぞれ、合成酵素はLasI、RhlI(ともにシュードモナス由来)と呼ばれる。このほかのAHL分子と、その合成酵素を以下に示した。これらのAHL分子は、アシル基のみが異なるため、一定の割合でLuxRと相互作用する(cross-talkする)。たとえば、OdHLとLuxRとのcross-talk(LasI-LuxR)は、OHHLとLuxRの場合(LuxI-LuxR)の1/100である.[http://www3.interscience.wiley.com/journal/119124142/abstract M.K Winson et al.:FEMS Microbiology Letters(1998)]
AHL分子の蓄積速度が一定ならば,luxI-LuxR/Pluxよりも20倍遅くスイッチが入ることとなる。
- クロストーク(その2)-LuxR/ Pluxの感度変異体を用いる場合-
LuxRの変異体(LuxR-G2E,LuxR-G2E-R67M)を用いると、3OC6HSL以外のAHLs(C10HSL, C6HSL, C8HSL, C12HSL,3OC12HSL)に対しても、receiverが活性化される。[http://www.nature.com/nbt/journal/v24/n6/abs/nbt1209.html C. H. Collins. et al.Nature Biotechnology.24, 708 - 712 (2006)]
Construction(From BioBrick)
More about Quorum Sensing
- [http://parts.mit.edu/registry/index.php/Featured_Parts:Cell-Cell-Signaling Cell-Cell-Signaling]
- [http://www.che.caltech.edu/groups/fha/quorum.html About Quorum sensing]
How Our System Works
| ホーム | メンバー紹介 | プロジェクト紹介 | 作成したDNAパーツ | ノート |
|---|
 "
"