Team:Chiba/Project
From 2008.igem.org
(→Signaling System) |
(→Plasmid Copynumber) |
||
| Line 118: | Line 118: | ||
[[Image:Receiver copy number chiba.jpg|frame|left|'''Fig.''' copynumber]]<br clear=all> | [[Image:Receiver copy number chiba.jpg|frame|left|'''Fig.''' copynumber]]<br clear=all> | ||
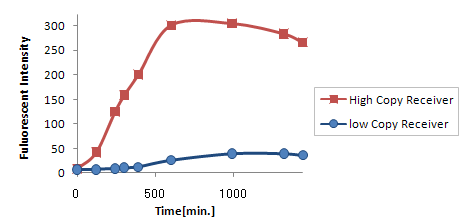
| - | + | レシーバーのコピーナンバーを変えることで、応答までの時間を変える<br> | |
コピーナンバーを変えれば、レシーバーによるLuxRの合成量は変化する<br> | コピーナンバーを変えれば、レシーバーによるLuxRの合成量は変化する<br> | ||
AHLを受け取るLuxRが変わるので応答閾値までの時間が変わるのだ<br> | AHLを受け取るLuxRが変わるので応答閾値までの時間が変わるのだ<br> | ||
Revision as of 15:14, 29 October 2008
| Home | The Team | The Project | Parts Submitted to the Registry | Reference | Notebook | Acknowledgements |
|---|
Abstract
E.coli time manager
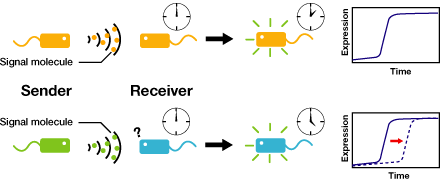
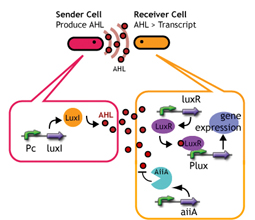
We are constructing delay switches to control/preset the timing of target gene expression. Our project uses two classes of bacteria: senders and receivers. Senders produce signaling molecules, and receivers are activated only after a particular concentration of this molecule is reached. The combinatorial use of senders/receivers allows us to make a‘switching consortium’which activates different genes at the preset times.
As signaling molecules, we utilize molecules associated with Quorum sensing, a phenomenon that allows bacteria to communicate with each other. Although different quorum sensing species have slightly different signaling molecules, these molecules are not completely specific to their hosts and cross-species reactivity is observed (1),(2). Communication using non-endogenous molecules is less sensitive than the original, and requires a higher signal concentration to take effect. This reduced sensitivity results in the slower activation of receivers, thus creating a system in which different receivers are activated after different amounts of time following signaling molecule release.
Introduction
Many electronic devices we use in our daily lives have the ability to keep track of time. For example, a VCR is able to record a TV program at a pre-set time, and a microwave automatically stops heating after a set amount of time. automatically stop heating when the right time comes. Using these temporal pre-programming functions, we have been liberated from either staying up late to watch a European soccer game or from worrying about our popcorn being burned black while yelling and shouting to the match we have videotaped. In this way, the timer function has revolutionized our lifestyle.
We thought the same applies to the biotechnology; we would like to freely implement the 'timer switches” to various biological functions, preferably both independently and in parallel format. These “functions” include sensors, synthesizers, or degraders of bioactive compounds/ materials, transportation and secretion machineries, communications, getting/ sticking together, proliferation and cell death. If successful, we will be able to program exceedingly more complex complex behaviors in cellular systems.
As one of the thousands of possible applications, we are trying to construct temporal imaging system using E. coli 'ink's that differ not in color but the 'timing' of coloration (fig). Over time, parts of images (or characters) are getting visible one by one, making animated message/ picture. In the end, the last ink get colorized, covering the entire image. Such system should be useful as a sort of secured communication board: we can convey our message to those who know the exact moment they should take a look. After a while, all the massage is gone.
Project Details
私たちは,2種類のbacteriaからなる Switching consortium によって,水時計型のtime delay装置をつくる(more about Water clock-wikipedia.en) 。 まず,Sender細胞が,ゆっくりとシグナルを合成する。それは次第に系に蓄積する。Receiverの応答感度に達した時,Receiverは遺伝子機能を起動するわけである(図説)。合成されたAHLが応答閾値に達するまでの時間を変えることで、遺伝子発現時間を調整することができる。そしてそのtime delay(preset time)は,sender側のmessage蓄積速度と,receiverの応答感度を調節すれば,任意に設定できる。
Signaling System
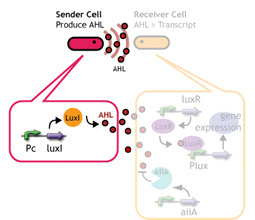
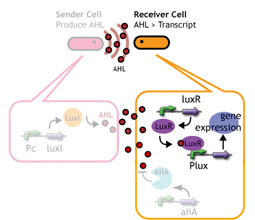
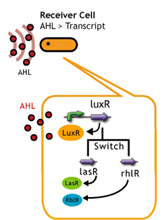
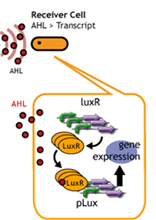
細胞間コミュニケーションであるクオラムセンシングを利用する。そもそもクオラムセンシングは,自分たちの密度がある閾値が一定値を超えたときに起動するものである。 Sender:LuxI protein familyがシグナル分子であるAHLを合成する。この分子は細胞膜を自由に通過し,Neighbering cellsにも感知される。 Receiver:常時LuxR proteinを発現し,AHLを監視している。AHL濃度がある値を超えると結合し,Lux promoter下の遺伝子発現をONにする。その応答閾値はLuxRとAHLとの親和性に依存している(文献)。 (more about quorum sensing)
System Design
|
プランA: Cross-talk
|
プランB: Tuning
|
|
|
|
|
|
Experiments and Result
Sender Crosstalk
Signal Molecule Receiver Phase
English:
日本語:AHLを合成するSenderだけではなく、AHLを受け取る側のReceiverを変えれば、その応答時間を変えることができる。そこで私たちは、以下のいくつかの方法を考えた。
- 一種類のSender(AHL<--LuxI)に対して、由来生物の異なるレシーバタンパク質でそれを受信する.
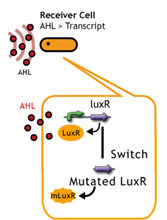
- レシーバータンパク質であるLuxRに変異を入れることで、AHLに対する応答感度を上下させること.
- レシーバーのコピーナンバーを変える.
Quorum-Sensing Crosstalkクオラムセンシングにおける、レシーバータンパクを変えてクロストークを起こさせる。 |
Plasmid Copynumberレシーバーのコピーナンバーを変えることで、応答までの時間を変える
|
|
LuxR mutant (Under construction)
レシーバータンパク質であるLuxRに変異を入れることで応答感度を上下させる(3),(4)
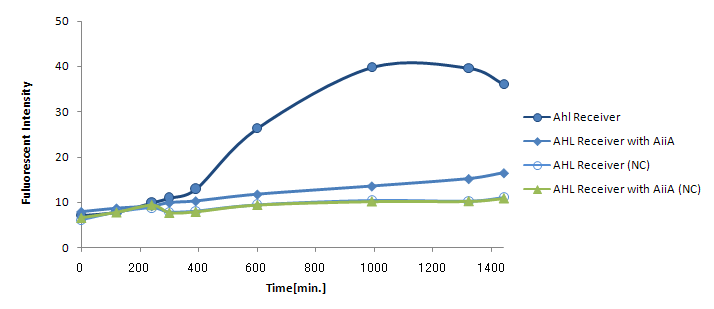
Signal Molecule Quencher
Design
|
more about AiiA Receiver Phase |
more about AiiA Receiver Experiment |
Demo Experiments
Demo ~Senders~
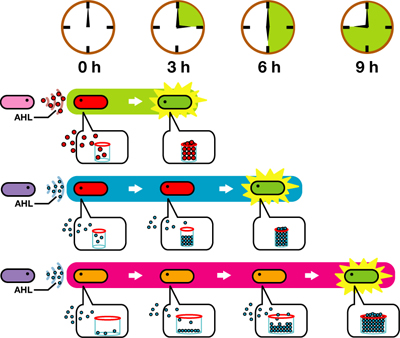
一番時差が見られたSender遺伝子のLuxIとLasIをつかってデモ実験を行った。
LuxIおよびLasIの遺伝子がそれぞれ組み込まれた大腸菌(XL10G)と、
LuxRの遺伝子が組み込まれた大腸菌(BW)を液体培養したものを 液量1:1で混ぜて、それらのGFPが発現するのを目視で観測した。
Results
結果は以下の通り。
1枚目の写真は開始点。2枚目はLuxIの部分だけGFPを肉眼(蛍光灯光)で確認。3枚目はLuxIとLasI両方を確認したものです。
(それぞれ0時間、4時間、8時間後の映像。液量が100μLと少ないため、今までの実験(1000μL)での結果よりもGFP発現に時間がかかった。・・・コレ書かない方がいいのかなあ。) --Yoshimi 11:04, 29 October 2008 (UTC)
-->more about Demo experiments detail
Demo ~Receivers~
English:
日本語:
固体培地中にセンダーBBa_S03623,(Ptet-LuxI) を混ぜ、固体培地表面にレシーバーのコロニーをN.Cフィルターで移す。
センダーの作るAHLは培地中を移動し、表面のレシーバーがAHLを一定濃度感知すればGFPを発現
する。一種のセンダーに対し、様々な種類のレシーバーを用いることで時間差が生じることを確認する。
用いるレシーバーは・・・
・シグナルを受け取るレシーバーを変える(クロストークの利用):LuxR,LasR,RhlR
・シグナル自体を分解するAiia を利用する
・レシーバーの遺伝子回路を含むプラスミドのコピーナンバーの変化
・レシーバータンパク質であるLuxRに変異を入れる
・確認の仕方
N.Cフィルターをはった個体培地を37°Cで培養し、時間(30min?)ごとにUVをあててGFPが見えるかチェックする。
香取
Results
--->more Demo experiments detail
Conclusion
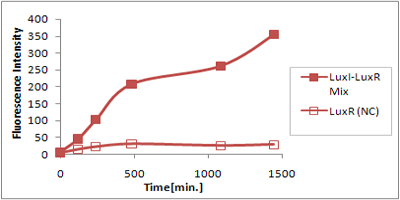
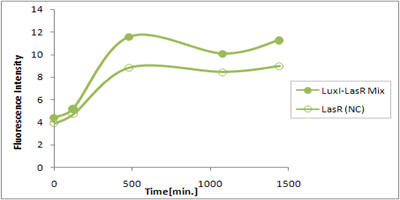
シグナル分子を3OC6HSLから3OC12HSLに変更することで、LuxRの応答時間を二時間遅らせることができた.
これらのSender実験とReceiver実験で時間差を作り出すことが出来たものを融合すれば、2時間以上の時間差の効果を期待することが出来る。
--Yoshimi 11:09, 29 October 2008 (UTC)
Future Work
- さらにLuxRのミューテーションやポジティブフィードバックループを利用することで、この時差を更に広げることが可能になり、発現時間のバリエーションがもっと増えるだろう。
Applications
時間の蓄積(クロストーク)を使うと…
セキュリティーシステム
GFPなどの目で確認できる文字(情報)を浮き上がらせてから、時間をおいて周りの菌にGFPを発現させる。
そうすとことによって、最初の文字(情報)を塗りつぶす。
もともと存在していた情報を消すことができる。
賞味期限装置
食品が傷む原因は細菌の活動が影響している。細菌の活動は温度に依存するので、温度変化を入力とする。
食品が傷む前にGFPを発現して私たちに知らせてくれる。
どれだけ時間変化が長くとも、短くとも私たちに知らせてくれる。
積分装置
消えてしまう入力(たとえばUV被ばく、汚染物質)をセンダーがAHLを出すことによって入力を記憶しておく。
AHLがたまり、危険量に達したことを知らせてくれる。
(杉山)
references
- M.K Winson et al.:Construction and analysis of luxCDABE-based plasmid sensors for investigating N-acyl homoserine lactone-mediated quorum sensing.FEMS Microbiology Letters 163 (1998) 185-192
- BBa_F2620:Specificity
- C. H. Collins.et al.:Directed evolution of Vibrio fischeri LuxR for increased sensitivity to a broad spectrum of acyl-homoserine lactones.Mol.Microbiol.2005.55(3).712–723
- B. Koch. et al.:The LuxR receptor: the sites of interaction with quorum-sensing signals and inhibitors.Microbiology 151 (2005),3589-3602
| Home | The Team | The Project | Parts Submitted to the Registry | Reference | Notebook | Acknowledgements |
|---|
 "
"