Team:KULeuven/Concreties
From 2008.igem.org
Result:
To have a dropdown for your own team copy over all of the content below. It might be needed to adjust a thing or two. Comments have been added throughout the code what must be changed.
Inspirational websites:
- [http://javascript-array.com/scripts/simple_drop_down_menu/ Most basic dropdown menu, with no submenu's]
- [http://jquery.com/ The javascript library used to add effects and optimise the dropdown]
- [http://www.tyssendesign.com.au/articles/css/centering-a-dropdown-menu/ How to center a ul list effectively]
The dropdown has been created and developed by the KULeuven team.
<html>
<style type="text/css">
#content {z-index:4;}
#ddwrapper * {z-index:8 !important;}
div#ddwrapper {
margin:0;
padding:0;
height:28px;
width:945px; /* change to adjust imperfections in width */
}
div#ddnav {
margin:0 auto; /* needed to center the dropdown */
padding:0;
top:5px;
/* width: 965px */
visibility:hidden; /* dropdown is hidden until properly initalised by javascript */
}
div#ddtoggle {
margin:0;
position:fixed;
right:2px;
top:15px;
height:10px;
width:10px;
z-index:100;
}
#ddnav ul {
display:table-row; /* works only for firefox, later adjusted by javascript for IE */
margin:0 auto;
padding:0;
}
#ddnav ul li {
display:table-cell; /* works only for firefox, later adjusted by javascript for IE */
list-style:none;
margin:0;
padding:0 !important;
border-right:1px solid #FFF; /* creates illusion of spacing between tabs */
}
#ddnav ul li:last-child{border-right:none;}
#ddnav a{
display:block;
margin:0;
padding:4px 14px; /* play with dimensions for size of tabs */
background-color:#075A90; /* background color for tabs */
color:#FFF !important; /* font color for text in tabs */
text-align:center; /* aligning for text in tabs */
text-decoration:none !important;
font:bold 10pt Trebuchet MS; /* font properties for text in tabs */
outline:0;
}
#ddnav ul li a:hover {background-color:#99CCFF;}/* background color for tabs on mouseover */
#ddnav li a:active {outline:none;} /* remove standard dotted border for links when clicked (IE) */
#ddnav li a:focus {-moz-outline-style:none;} /* remove standard dotted border for links when clicked (FF) */
#ddnav div {
display:none;
position:absolute;
width:9em;
background-color:#000; /* bug solution, do not change ! */
border:1px solid #5970B2; /* border color for dropdown menus */
opacity:0.9; /* transparancy of the dropdown menus (FF) */
filter:alpha(opacity=90); /* transparancy of the dropdown menus (IE) */
}
#ddnav div a {
display:block;
padding:5px 10px; /* play with dimensions of block element in dropdown menus */
position:relative;
font:normal 8pt arial; /* font properties for text in dropdown menus */
text-align:left; /* aligning of text in dropdown menus */
cursor:pointer;
}
#ddnav div a:hover, #ddnav span a:hover {color:#000 !important;} /* text color on mouseover */
#ddnav span div {
position:relative;
border:none;
border-bottom:2px groove #5970B2; /* separator for submenus, groove does not work in FF */
opacity:1.0; /* avoid stacking transparancy for submenus (FF) */
filter:alpha(opacity=100); /* avoid stacking transparancy for submenus (IE) */
}
/* may want to upload the following pictures to a new location */
.expand {background: url('https://static.igem.org/mediawiki/2008/e/ef/Icon-expand.png') no-repeat 95% 50%;}
.collapse {background: url('https://static.igem.org/mediawiki/2008/c/cd/Icon-collapse.png') no-repeat 95% 50%;}
.docked {background: #99ccff url("https://static.igem.org/mediawiki/2008/6/62/Ddnavundock.png") no-repeat 50% 50%;}
.undocked {background: #99ccff url("https://static.igem.org/mediawiki/2008/e/e4/Ddnavdock.png") no-repeat 50% 50%;}
</style>
<!-- IMPORTANT: save following script under a personalized webspace or download the library at www.jquery.com -->
<script type="text/javascript" src="http://student.kuleuven.be/~s0173901/wiki/js/jquery.js"></script>
<script type="text/javascript">
function ddnav() {
$('#ddnav ul li').hover(
function () {
$(this).find('div:first').css('display','block');},
function () {
$(this).find('div:first').css('display','none');}
);
}
function ddnavsub() {
$('#ddnav span > a').toggle(
function () {
$(this).removeClass("#ddnav expand").addClass("#ddnav collapse");
$(this).parent().find('div:first').slideDown('fast');
$(this).hover(
function (){$(this).css('background-color','#99AAFF');},
function (){$(this).css('background-color','#99AAFF');});},
function () {
$(this).removeClass("#ddnav collapse").addClass("#ddnav expand");
$(this).parent().find('div:first').css('display','none');
$(this).hover(
function (){$(this).css('background-color','#99CCFF');},
function (){$(this).css('background-color','#075A90');});}
).addClass("#ddnav expand");
}
/* If you wish to omit the docking feature, remove following function ddtoggle() */
function ddtoggle() {
$('#ddtoggle').toggle(
function () {
$(this).removeClass('undocked').addClass('docked');
$('#ddnav').css('position','fixed');},
function () {
$(this).removeClass('docked').addClass('undocked');
$('#ddnav').css('position','static');}
);
}
function ddalign() {
var _windowWidth = $(window).width();
var _leftOffset = (_windowWidth - 965)/2;
$('div#ddnav').css('left',_leftOffset);
}
function ddmsie() {
$('#ddnav a').hover(
function () {$(this).css('background-color','#99ccff');},
function () {$(this).css('background-color','#075a90');}
);
/* toggle doesn't work yet */
$('#ddtoggle').css('display','none');
$('#ddnav ul').css('display','inline-block');
$('#ddnav ul li').css('display','inline');
$('#ddnav ul li').css('position','relative');
$('#ddnav ul li>a').css('display','inline-block');
$('#ddnav ul li>a').css('margin-right','-4px');
$('#ddnav div').css('left','0');
$('#ddnav div').css('top','100%');
$('#ddnav span div').css('top','0');
}
function ddmozilla() {
ddtoggle();
$(window).bind('resize', function() {ddalign();});
}
$(function () {
ddnav();
ddnavsub();
if(jQuery.browser.msie) ddmsie();
if(jQuery.browser.mozilla) ddmozilla();
$('#ddnav').css('visibility','visible');
});
</script>
<!-- If you wish to omit the docking feature omit following line (div with id ddtoggle) -->
<div id="ddtoggle" class="undocked"></div>
<div id="ddwrapper">
<!-- Here the actual links are defined, simply replace with your own links in the appropriate sections -->
<div id="ddnav" align="center">
<ul>
<li>
<a href="https://2008.igem.org/Team:KULeuven">Home</a>
</li>
<li>
<a>The Team</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Team/LabsandGroups">Research Labs and Groups</a>
<a href="https://2008.igem.org/Team:KULeuven/Team/Students">Students</a>
<a href="https://2008.igem.org/Team:KULeuven/Team/Instructors">Instructors</a>
<a href="https://2008.igem.org/Team:KULeuven/Team/Advisors">Advisors</a>
<a href="https://2008.igem.org/Team:KULeuven/Team/Pictures">Pictures</a>
</div>
</li>
<li>
<a>The Project</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Project">Summary</a>
<span>
<a>Components</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Project/Input">Input</a>
<a href="https://2008.igem.org/Team:KULeuven/Project/Output">Output</a>
<a href="https://2008.igem.org/Team:KULeuven/Project/Filter">Filter</a>
<a href="https://2008.igem.org/Team:KULeuven/Project/Inverter">Invertimer</a>
<a href="https://2008.igem.org/Team:KULeuven/Project/Reset">Reset</a>
<a href="https://2008.igem.org/Team:KULeuven/Project/CellDeath">Cell Death</a>
<a href="https://2008.igem.org/Team:KULeuven/Project/Memory">Memory</a>
</div>
</span>
<a href="https://2008.igem.org/Team:KULeuven/Evaluation">End Evaluation</a>
<a href="https://2008.igem.org/Team:KULeuven/Literature">Literature</a>
<a href="https://2008.igem.org/Team:KULeuven/Brainstorm">Brainstorm</a>
</div>
</li>
<li>
<a>Ethics</a>
<div>
</div>
</li>
<li>
<a>Submitted Parts</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Parts">Listing</a>
<a href="http://partsregistry.org/cgi/partsdb/pgroup.cgi?pgroup=iGEM2008&group=KULeuven">Sandbox</a>
</div>
</li>
<li>
<a>Modeling</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Model/Overview">Overview</a>
<a href="https://2008.igem.org/Team:KULeuven/Model/KineticConstants">Kinetic Constants</a>
<span>
<a>Components</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Model/Output">Output</a>
<a href="https://2008.igem.org/Team:KULeuven/Model/Filter">Filter</a>
<a href="https://2008.igem.org/Team:KULeuven/Model/Inverter">Invertimer</a>
<a href="https://2008.igem.org/Team:KULeuven/Model/Reset">Reset</a>
<a href="https://2008.igem.org/Team:KULeuven/Model/CellDeath">Cell Death</a>
<a href="https://2008.igem.org/Team:KULeuven/Model/Memory">Memory</a>
</div>
</span>
<a href="https://2008.igem.org/Team:KULeuven/Model/FullModel">Full Model</a>
<a href="https://2008.igem.org/Team:KULeuven/Model/Sensitivity">Sensitivity Analysis</a>
<a href="https://2008.igem.org/Team:KULeuven/Model/MultiCell">Multi-cell Model</a>
<a href="https://2008.igem.org/Team:KULeuven/Model/Diffusion">Diffusion</a>
</div>
</li>
<li>
<a>Data Analysis</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Data/Overview">Overview</a>
<span>
<a>New Parts</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Data/GFP">GFP (LVA-tag)</a>
<a href="https://2008.igem.org/Team:KULeuven/Data/T7">T7 (UmuD-tag)</a>
<a href="https://2008.igem.org/Team:KULeuven/Data/Antisense">Antisense LuxI</a>
<a href="https://2008.igem.org/Team:KULeuven/Data/ccdB">Celldeath (ccdB)</a>
<a href="https://2008.igem.org/Team:KULeuven/Data/HybridProm">Hybrid Promotor</a>
</div>
</span>
<span>
<a>Components</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Data/Input">Input</a>
<a href="https://2008.igem.org/Team:KULeuven/Data/Output">Output</a>
<a href="https://2008.igem.org/Team:KULeuven/Data/Filter">Filter</a>
<a href="https://2008.igem.org/Team:KULeuven/Data/Inverter">Invertimer</a>
<a href="https://2008.igem.org/Team:KULeuven/Data/Reset">Reset</a>
<a href="https://2008.igem.org/Team:KULeuven/Data/CellDeath">Cell Death</a>
<a href="https://2008.igem.org/Team:KULeuven/Data/Memory">Memory</a>
</div>
</span>
<a href="https://2008.igem.org/Team:KULeuven/Data/FullModel">Full Model</a>
</div>
</li>
<li>
<a>Software</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Software/MultiCell">Multi-cell Toolbox</a>
<a href="https://2008.igem.org/Team:KULeuven/Software/Simbiology2LaTeX">Simbiology2LaTeX Toolbox</a>
</div>
</li>
<li>
<a>Notebook</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Meeting_Calendar">Calendar</a>
<a href="https://2008.igem.org/Team:KULeuven/SummerHolidays">Summer Holidays</a>
<span>
<a>Reports</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Meeting Reports">Daily</a>
<a href="https://2008.igem.org/Team:KULeuven/Weekly Meetings">Weekly</a>
</div>
</span>
<span>
<a>Lab Data</a>
<div>
<a href="https://2008.igem.org/Team:KULeuven/Freezer">Freezer</a>
<a href="https://2008.igem.org/Team:KULeuven/Primers">Primers</a>
<a href="https://2008.igem.org/Team:KULeuven/Ligation">Ligation</a>
</div>
</span>
<a href="https://2008.igem.org/Team:KULeuven/Tools">Tools</a>
<a href="https://2008.igem.org/Team:KULeuven/Press">Press</a>
<a href="https://2008.igem.org/Team:KULeuven/Guestbook">Guestbook</a>
</div>
</li>
</ul>
</div>
</div>
</html>
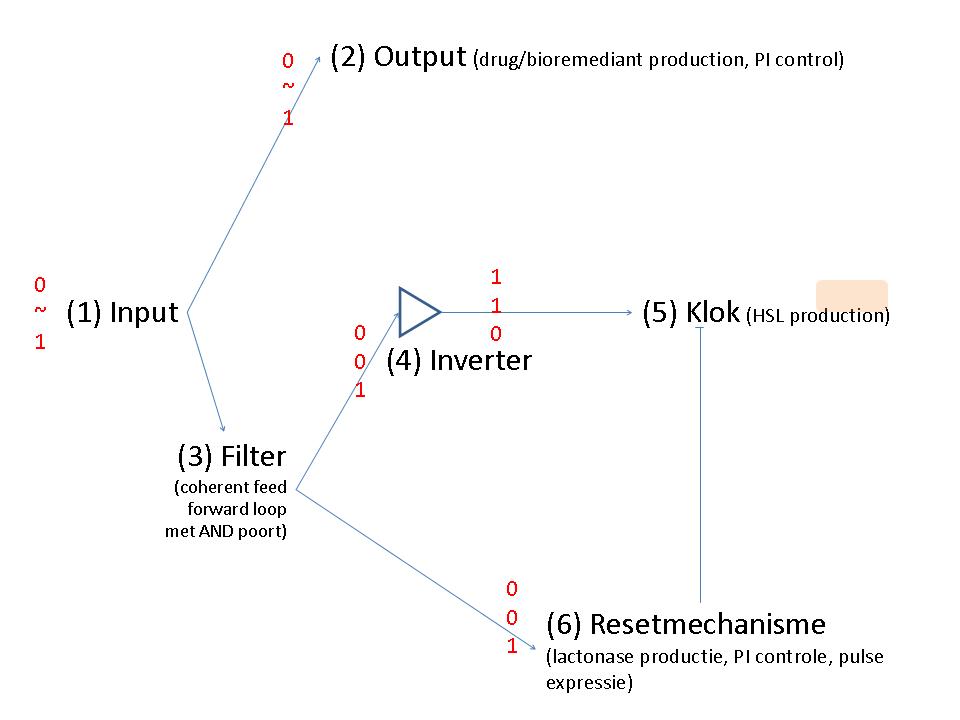
Op deze pagina verzamelen we concrete invullingen omtrent het project. De bedoeling is niet meer om over conceptuele dingen na te denken, maar om bijvoorbeeld op zoek te gaan naar analogen met vorige projecten, bruikbare BioBricks enzoverder. Voorlopig gooien we het nog niet onmiddellijk op de voorgrond, tot we meer voet in de aarde hebben omtrent haalbaarheid en zo.
Contents |
Project PI
Input
The input could be abstracted by using an easily controllable light flash.
OmpR- en 660nm- gereguleerd
The system of the Austin 2004 iGEM team cannot be used (or is better not used), because the light flash inhibits the promoter, where we need a light flash that activates a promoter.
- Aparently, OmpR-P has a different effect on ompC and ompR (see [http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=106865 this paper]). In Levskaya et al, they used the promoter of ompC BBa_R0082, which would indeed not be useful for us since a flash of light would inhibit autophosphorylation of the sensor, and thus OmpR and hence turn the system off. Maybe we can use the promoter of ompF? The team of [http://openwetware.org/wiki/IGEM:Melbourne/2008/BioClock/SPModel Melbourne] is using the promoter of ompF, that's where I got the inspiration to use the flash light as a dummy input. Maybe other dummy inputs are possible? Light has the advantage of being easy to manipulate of course. -Ingethijs 14:39, 3 July 2008 (UTC)
- Indeed, OmpF is activated by OmpR (without phosphate) and is repressed by OmpR-P. See [http://www.jbc.org/cgi/reprint/269/17/12559 this paper]. The OmpF promoter is available as BioBrick BBa_R0084. This could be a feasible mechanism for dummy input. - Bmoeyaert
Ik zou in dit systeem dan volgende modules voorstellen:
- BBa_R0040: medium strength promoter
- BBa_B0034: RBS
- BBa_I15008: maakt biliverdine IXalfa uit de heem-group
- BBa_B0034: RBS
- BBa_I15009: maakt fycobilicyanine uit biliverdine IXalfa
- BBa_B0010: terminator
- BBa_B0012: terminator
- BBa_R0040: medium strength promoter
- BBa_B0034: RBS
- BBa_I15010: maakt een Cph1 licht receptor/EnvZ fusieproteïne, een kinase dat wordt gedeactiveerd bij 660nm, bij afwezigheid van 660nm fosforyleert het OmpR
- BBa_B0010: terminator
- BBa_B0012: terminator
- BBa_R0084: OmpF promoter, wordt geactiveerd door WEINIG OmpR-P (zoals bij deactivatie van EnvZ door 660nm) en wordt geactiveerd door veel OmpR-P (zoals bij afwezigheid van 660nm, wanneer EnvZ actief is)
- BBa_B0034: RBS
- BBa_C0051: snel degraderende cI lambda repressor
- BBa_B0010: terminator
- BBa_B0012: terminator
- Deze modules zorgen er in het kort voor dat bij 660nm licht, er cI lambda repressor wordt aangemaakt. Deze kan als signaal dienen voor bijvoorbeeld de output, dankzij BBa_I12007, een door cI geïnduceerde promoter.
- We moeten er met dit systeem voor opletten dat we nergens anders een cI-afhankelijke promoter gebruiken, die niet rechtstreeks afhankelijk is van het inputsignaal.
- De gebruikte bacteriële stam mag het EnvZ-gen niet bevatten.
Andere mogelijkheden
- There is a BioBrick (BBa_I765001) that is induced by UV-light. This would be perfect: only UV light could than trigger our system. This BioBrick is not confirmed to work, and there is very little information availabla about it. I doubt its proper working, however.
- Harvard 2005 also did something with UV. It involved the lambda cI repressor, and resulted in UV light switching something on. I do not (yet) understand how it works. See http://openwetware.org/images/8/86/Doucette_et_al._2006.pdf.
Output
Als output is GFP waarschijnlijk het handigste. Dankzij een C-terminale tag ([http://aem.asm.org/cgi/content/full/64/6/2240?view=long&pmid=9603842 RPAADNYALAA]) kan de half-life verminderd worden tot 40min tot enkele uren. Ik stel volgende output module voor:
- BBa_I12007: lambda Prm promoter, activatie door cI, geen repressie door aanwezigheid mutatie in OR3
- BBa_B0034: RBS
- BBa_XXXXXX: snel degraderend GFP dankzij [http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=106306 RPAADNYALAA] aan C-terminaal uiteinde
- BBa_B0010: terminator
- BBa_B0012: terminator
Filter
This is what I found (up to now).
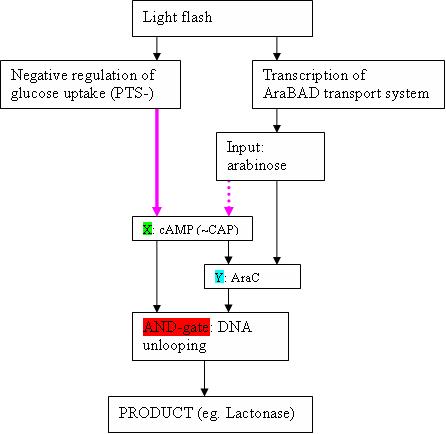
- In the paper of Shen-Orr , the example of the feedforward loop with AND-gate is the well-studied Ara-operon (This can occur when X and Y act in an ‘AND-gate’−like manner to control operon Z, as is the case in the araBAD operon in the arabinose feedforward loop)
- Is dit het enige voorbeeld dat ze aanhalen in shen-orr? misschien is dit systeem nogal moeilijk omvormbaar naar een specifieke filter vermits cAMP nogal een belangrijke rol speelt in de cel – het idee was dan ook om de input van X te veranderen in een induceerbare promoter naar keuze, bijv een OmpR afhankelijke als we het systeen van levskaya met de lichtpuls gebruiken -- IT
- The input signal is of course arabinose. We will need AraF(GH?) to transport it efficiently from the medium into the cell.
- What is depicted (see figure) as X, is cAMP (+ CAP)
- Y is AraC
- The AND gate is the “unlooping” of the DNA strand in de Ara operon. This happens only in the presence of both AraC bound to arabinose, and CAP-cAMP
- Product Z are in a WT Ara operon the AraBAD proteïns, but can of course be changed to eg. lactonase. The araBAD genes are located downstream the pBAD promoter, which is activated by the “DNA unlooping”.
- Kan Z ook een tussenproduct zijn dat enerzijds de inverter ingaat richting HSL productie en anderzijds, na het ribolock-key systeem, lactonase productie aanstuurt -- IT
Note that Y is also to be stimulated by arabinose, so the scheme is actually a bit more complex than the easy scheme depicted in the Shen-Orr paper
Some problems
- I cannot find literature about the dotted purple arrow: is it überhaupt existing? The article of Shen-Orr suggests it does.
- cAMP concentration is depressed by glucose, so maybe the purple arrow means that the concentration of glucose is negligible compared to arabinose. This might however not be good enough.
- What could be done, is making sure that not only AraBAD is made upon (eg) flashing with a light (Austin iGEM 2004), but also making a repressor for the phosphotransferase system of glucose transport. However, then, the pink purple arrow should be the full (non-dotted) pink one.
- I did not (yet) find the BioBricks of the Austin 2004 team for the input signal.
- I believe they can be found here: http://partsregistry.org/Featured_Parts:Light_Sensor -- IT
- Adenylate cyclase inhibition is coupled to the flux of glucose through the glucose PEP:phosphotransferase system . This means that external glucose concentration regulates cAMP production. This is I think a major problem: how do we arrange the system so that it becomes dependent on cytoplasmatic factors?
- Vandaar dat ik hoop dat er nog een ander systeem in E.coli gekend is dat dit netwerkmotief heeft, daar waren we een beetje vanuit gegaan... -- IT
- Ik denk dat het systeem zoals hierboven uitgelegd moet lukken. Prof. Winderickx vertelde dat cAMP zowel afhankelijk is van intra- als extracellulaire glucose-concentratie. Dat lost het probleem hier geformuleerd op. Daarenboven klinkt de paper die hieronder geciteerd staat zeer veelbelovend, zij hebben immers al uitvoerig het ara-operon in min of meer hetzelfde perspectief als hier beschreven bestudeerd. Bmoeyaert
- If cAMP is not degraded fast enough, could cyclic nucleotide phosphodiesterases catalyse this process of degradation?
Interesting reads
- http://www.weizmann.ac.il/mcb/UriAlon/Papers/The%20Coherent%20Feedforward%20Loop%20Serves%20as%20a%20Sign-sensitive%20Delay%20Element%20in%20Transcription%20Networks.pdf
Inverter
Als inverter zouden bijvoorbeeld BBa_Q04400 of BBa_Q04121 kunnen gebruikt worden. Deze zijn immers:
- onafhankelijk van cI
- onafhankelijk van eender welke component van het Ara Operon (in zoverre we dat als filter gebruiken)
Klok
Resetmechanisme
pulsexpressie van lactonase:
van Johan Robben - 7 juli:
een pulsgenerator met beschikbare BioBricks
De regulatorische delen zijn zo gekozen dat ze niet interfereren met andere genen in coli. Voor zover ik kan zien is het gebruik van een tandem-promoter een nieuwigheid, en misschien op zich al het proberen waard. 't Zou nog leuker zijn natuurlijk als ze verbeteringen kunnen aanbrengen.
Project D
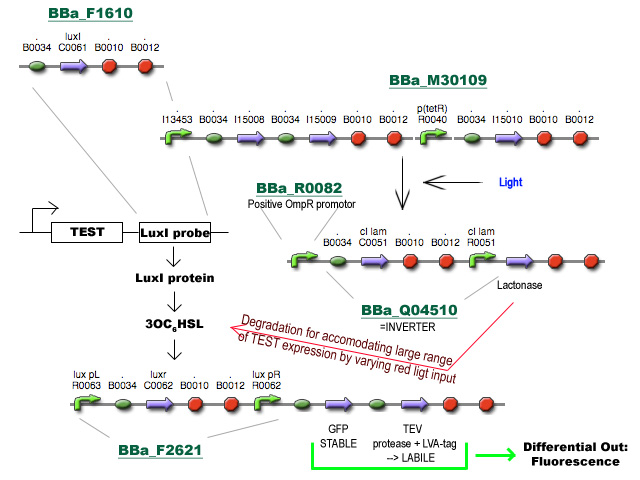
Zoals gevraagd, een concrete blik op Project D:
Ik ben vertrokken van het voorstel van Prof. Robben en heb nog een stukje toegevoegd om tegemoet te komen aan de brede range van expressieniveaus in de cel. Het gebruik van HSL synthese en degradatie (vanuit andere voorstellen) zorgt respectievelijk voor amplificatie van het signaal van differentiele regulatie en voor het verhogen van de dynamic range van expressieniveaus die het systeem aankan via belichting met rood licht op verschillende intensiteiten. Enfin, I had fun. Gooi er maar vragen/opmerkingen onder :)
(Begin linksboven) -Jonas 17:13, 5 July 2008 (UTC)
- Ik zie dat je gebruikt maakt van de pBAD promoter en de OmpR regulator. Indien we dit willen integreren met het project hierboven, zal ofwel deze stap, ofwel de input andere vorm moeten aannemen, want beide werken met de OmpR regulator. - Bmoeyaert
- Kan je eventueel een stroomschema maken van wat er juist "gebeurt", want ik kan het schema maar moeilijk volgen (en ik denk dat ik de enige niet ben). - Bmoeyaert
Ik zal in de loop van de dag een beetje een flow-chart proberen te maken. Zal hier voorlopig nu een korte beschrijving proberen te geven.
- BBa_F1610 fungeert als probe om het expressieniveau van een TEST gen te volgen. Deze BioBrick wordt achter het TEST gen geïnsereerd (vóór de transcriptie terminators, misschien zijn die van BBa_F1610 niet eens nodig) en zal vertaald worden tot LuxI proteine.
- LuxI produceert 3-oxohexanoyl-homoserine lactone (3OC6HSL) wat gesensed wordt via LuxR, geproduceerd door BioBrick BBa_F2621.
- De aanwezigheid van LuxR met gebonden 3OC6HSL fungeert als transcriptie activator vanaf de Lux pR promotor en initieert transcriptie van een stabiel en snel vouwend Green Fluorescent Protein met TEV herkenningssequentie en een labiel TEV protease. (met de sterkte van de Ribosome Binding Sites zal nog gespeeld moeten worden)
- TEV degradeert het GFP en zorgt voor een differentieel fluorescent signaal dat uitgelezen kan worden.
- Het stuk rechtsboven is basically een regelbare cascade die uitmondt in de productie van Lactonase wat de 3OC6HSL geproduceerd door LuxI afbreekt. Door de hoeveelheid lactonase onder fijne gegradeerde regulatie te plaatsen (hier nu bvb licht genomen) wordt de range van mogelijke expressieniveaus waarmee het systeem overweg kan uitgebreid.
- Al wat BBa_M30109 doet is lichtsensor produceren onder controle van pBAD en pTet. In het donker zorgt deze BioBrick voor transcriptie vanaf de OmpR promotor(BBa_R0082). Dit signaal gaat door de inverter BBa_Q04510 die output geeft via lactonase. In het donker wordt er dus geen extra lactonase tot expressie gebracht.
- Als het gebruik van pBAD en pOmpR lastig is kan lactonase simpelweg onder pLac (BBa_I14032) controle geplaatst worden, waar de hoeveelheid lactonase gecontroleerd kan worden door het toevoegen van IPTG. Heel het stuk rechtsboven kan dus eigenlijk ook door 1 enkele part vervangen worden onder de vorm van pLac-RBS-lactonase-STOP ;) -Jonas
PS: If the D is to be integrated in the PI project (mind the pun :P ) after the output-gene for example, the lactonase will have to go and a different HSL synthesis-sensor system will have to be used (examples are in the registry, minimal cross-reaction is a requirement)
 "
"