Team:KULeuven/Concreties
From 2008.igem.org
m |
|||
| (7 intermediate revisions not shown) | |||
| Line 1: | Line 1: | ||
| - | {{:Team:KULeuven/Tools/ | + | {{:Team:KULeuven/Tools/Styling}} |
| + | {{:Team:KULeuven/Tools/Scripting}} | ||
| + | {{:Team:KULeuven/Tools/Header}} | ||
| - | + | = Project = | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | = Project | + | |
[[Image:idea_3.jpg|500px|center]] | [[Image:idea_3.jpg|500px|center]] | ||
| - | ==Input== | + | == Input == |
| + | [[Image:input.png|500px|center]] | ||
| - | |||
| - | === | + | == Output == |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | [[Image:output.png|500px|center]] | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | == Filter == | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
| - | |||
[[Image:SchemaFilter2.png|500px|center]] | [[Image:SchemaFilter2.png|500px|center]] | ||
| - | ==Inverter== | + | == Inverter == |
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | + | ||
| - | == | + | == Clock == |
| - | == | + | == Reset mechanism == |
pulsexpressie van lactonase: | pulsexpressie van lactonase: | ||
| Line 89: | Line 40: | ||
Het enige waar ik mee zit is de efficientie van de transcriptie van cI vanaf pTet wanneer er cI op P<sub>R</sub> gebonden is. Dit kan misschien doorlezen van het RNA polymerase belemmeren maar zal waarschijnlijk wel ok zijn aangezien de concentratie cI voldoende laag gehouden wordt. In ieder geval, me like -[[User:Zeunas|Jonas]] | Het enige waar ik mee zit is de efficientie van de transcriptie van cI vanaf pTet wanneer er cI op P<sub>R</sub> gebonden is. Dit kan misschien doorlezen van het RNA polymerase belemmeren maar zal waarschijnlijk wel ok zijn aangezien de concentratie cI voldoende laag gehouden wordt. In ieder geval, me like -[[User:Zeunas|Jonas]] | ||
| - | = Project D = | + | == Project D == |
Zoals gevraagd, een concrete blik op Project D: | Zoals gevraagd, een concrete blik op Project D: | ||
Latest revision as of 14:53, 3 October 2008
Contents |
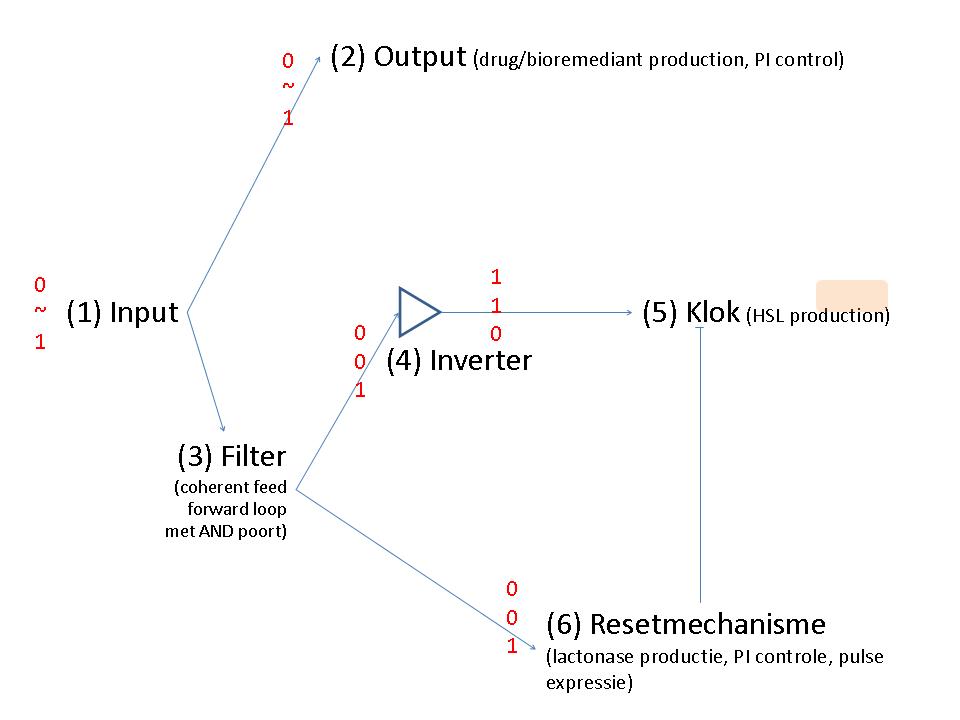
Project
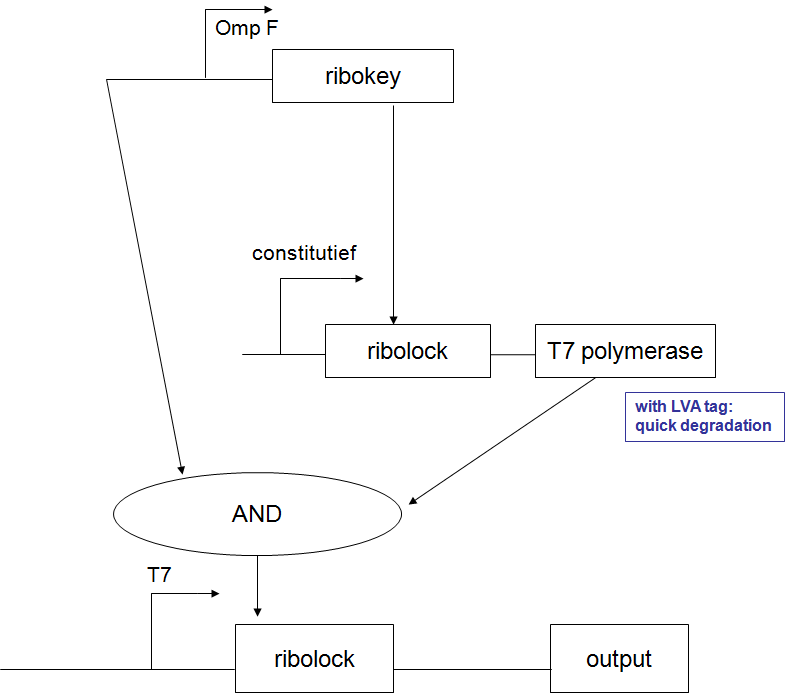
Input
Output
Filter
Inverter
Clock
Reset mechanism
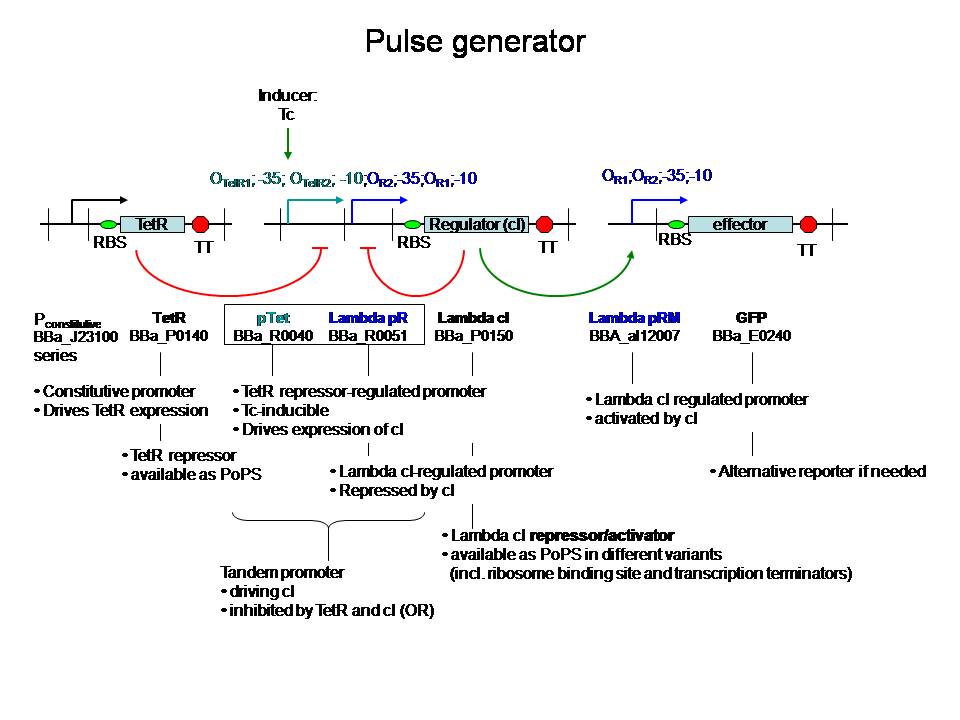
pulsexpressie van lactonase:
van Johan Robben - 7 juli:
een pulsgenerator met beschikbare BioBricks
De regulatorische delen zijn zo gekozen dat ze niet interfereren met andere genen in coli. Voor zover ik kan zien is het gebruik van een tandem-promoter een nieuwigheid, en misschien op zich al het proberen waard. 't Zou nog leuker zijn natuurlijk als ze verbeteringen kunnen aanbrengen.
Fijn hoe het hier in de details zit. Het gebruik van de partiele PR en PRM met dus omgekeerde OR1 en OR2 waardoor cI productie gerepressed wordt voordat de concentratie hoog genoeg wordt om effector transcriptie te initieren. Het enige waar ik mee zit is de efficientie van de transcriptie van cI vanaf pTet wanneer er cI op PR gebonden is. Dit kan misschien doorlezen van het RNA polymerase belemmeren maar zal waarschijnlijk wel ok zijn aangezien de concentratie cI voldoende laag gehouden wordt. In ieder geval, me like -Jonas
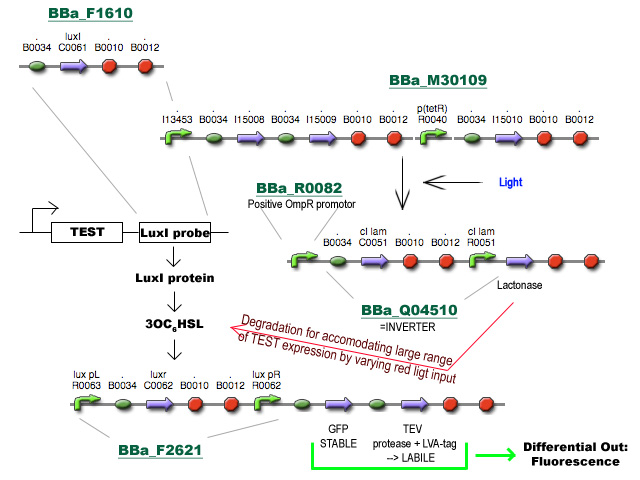
Project D
Zoals gevraagd, een concrete blik op Project D:
Ik ben vertrokken van het voorstel van Prof. Robben en heb nog een stukje toegevoegd om tegemoet te komen aan de brede range van expressieniveaus in de cel. Het gebruik van HSL synthese en degradatie (vanuit andere voorstellen) zorgt respectievelijk voor amplificatie van het signaal van differentiele regulatie en voor het verhogen van de dynamic range van expressieniveaus die het systeem aankan via belichting met rood licht op verschillende intensiteiten. Enfin, I had fun. Gooi er maar vragen/opmerkingen onder :)
(Begin linksboven) -Jonas 17:13, 5 July 2008 (UTC)
- Ik zie dat je gebruikt maakt van de pBAD promoter en de OmpR regulator. Indien we dit willen integreren met het project hierboven, zal ofwel deze stap, ofwel de input andere vorm moeten aannemen, want beide werken met de OmpR regulator. - Bmoeyaert
- Kan je eventueel een stroomschema maken van wat er juist "gebeurt", want ik kan het schema maar moeilijk volgen (en ik denk dat ik de enige niet ben). - Bmoeyaert
Ik zal in de loop van de dag een beetje een flow-chart proberen te maken. Zal hier voorlopig nu een korte beschrijving proberen te geven.
- BBa_F1610 fungeert als probe om het expressieniveau van een TEST gen te volgen. Deze BioBrick wordt achter het TEST gen geïnsereerd (vóór de transcriptie terminators, misschien zijn die van BBa_F1610 niet eens nodig) en zal vertaald worden tot LuxI proteine.
- LuxI produceert 3-oxohexanoyl-homoserine lactone (3OC6HSL) wat gesensed wordt via LuxR, geproduceerd door BioBrick BBa_F2621.
- De aanwezigheid van LuxR met gebonden 3OC6HSL fungeert als transcriptie activator vanaf de Lux pR promotor en initieert transcriptie van een stabiel en snel vouwend Green Fluorescent Protein met TEV herkenningssequentie en een labiel TEV protease. (met de sterkte van de Ribosome Binding Sites zal nog gespeeld moeten worden)
- TEV degradeert het GFP en zorgt voor een differentieel fluorescent signaal dat uitgelezen kan worden.
- Het stuk rechtsboven is basically een regelbare cascade die uitmondt in de productie van Lactonase wat de 3OC6HSL geproduceerd door LuxI afbreekt. Door de hoeveelheid lactonase onder fijne gegradeerde regulatie te plaatsen (hier nu bvb licht genomen) wordt de range van mogelijke expressieniveaus waarmee het systeem overweg kan uitgebreid.
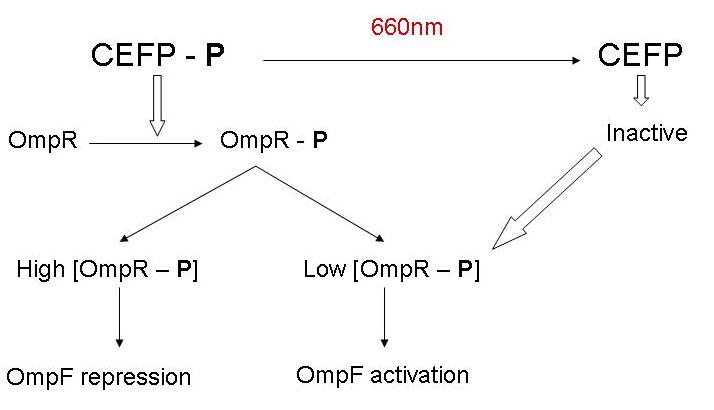
- Al wat BBa_M30109 doet is lichtsensor produceren onder controle van pBAD en pTet. In het donker zorgt deze BioBrick voor transcriptie vanaf de OmpR promotor(BBa_R0082). Dit signaal gaat door de inverter BBa_Q04510 die output geeft via lactonase. In het donker wordt er dus geen extra lactonase tot expressie gebracht.
- Als het gebruik van pBAD en pOmpR lastig is kan lactonase simpelweg onder pLac (BBa_I14032) controle geplaatst worden, waar de hoeveelheid lactonase gecontroleerd kan worden door het toevoegen van IPTG. Heel het stuk rechtsboven kan dus eigenlijk ook door 1 enkele part vervangen worden onder de vorm van pLac-RBS-lactonase-STOP ;) -Jonas
PS: If the D is to be integrated in the PI project (mind the pun :P ) after the output-gene for example, the lactonase will have to go and a different HSL synthesis-sensor system will have to be used (examples are in the registry, minimal cross-reaction is a requirement)
 "
"